ゲノム遺伝解析学
ゲノム遺伝解析学 (Plant Genome Dynamics Analysis)
転移因子を利用した作物ゲノムの遺伝解析とその応用
教員
 | 准教授: 門田 有希 Assoc. Prof. Dr. Yuki MONDEN E-mail: y_monden@(@以下はokayama-u.ac.jp を付けてください。) 専門分野: 植物遺伝育種学 |
主な研究テーマ
私たちの研究室では,“レトロトランスポゾン”という,動く遺伝子に注目して研究を行っています.このレトロトランスポゾンというのは,自分のコピー配列を作って増えている,非常に興味深いDNA配列です.私たちは,このレトロトランスポゾンを利用し,さまざまな遺伝解析を行っています.また,近年急速に技術開発の進んだ次世代シーケンス(NGS-Next Generation Sequencing)も積極的に取り入れ,効率的に大規模な遺伝解析を行っています.
キーワード:レトロトランスポゾン,次世代シーケンス,倍数性,サツマイモ,DNAマーカー,連鎖解析
遺伝解析に有用なファミリーのスクリーニング
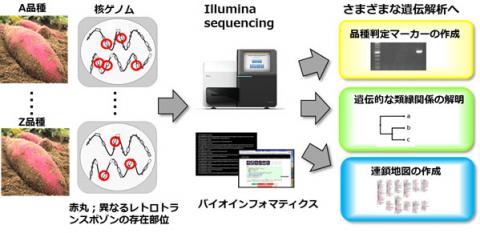
生物のゲノム中には,非常にたくさんのレトロトランスポゾンファミリー(種類のこと)が存在しています.それらファミリーのほとんどは,今は動いていません。が,未だに動いているファミリーもごく少数,存在しています.そして、それら動くファミリーの染色体上の存在箇所は農作物の品種間で大きく異なります.そのため,存在箇所の違いは,DNA配列上の目印(DNAマーカー)となり,遺伝解析や品種判定のツールとして使うことができます.私たちは,NGSを利用した大規模な解析により,このような遺伝解析に有用なファミリーを効率的に見つけ出す方法を開発しています.
レトロトランスポゾンを利用したジェノタイピング
レトロトランスポゾンの存在箇所の違いを利用し,さまざまな遺伝解析を行っています.その際,多数の品種(サンプル)に関して,多数の挿入部位を効率的に同定するため,NGSを利用しています.それにより,(i) 農作物品種の保護,食の安心・安全に向けた,品種判定マーカーの開発,(ii) 栽培進化の解明などに向けた,品種間の類縁関係の解明,(iii)農業上有用な遺伝子を同定するための連鎖地図の作成など,さまざまな遺伝解析に取り組んでいます.対象の農作物種は,サツマイモ,イチゴ,ダイズ,リンゴ,カンキツなどです.
有用遺伝子同定に向けた連鎖地図の作成
|
農業上有用な遺伝子(病害抵抗性,機能性成分付与など)を同定するため,連鎖地図を作成しています.異なる特徴をもつ品種(例えば,病害に強い,弱いなど)を交配し,後代集団を育成します.それを用いて,DNAマーカーによる遺伝子型解析,さらに病気に強いか弱いかという形質を調査し,連鎖解析を行います.これにより,遺伝子の同定,あるいは選抜マーカーの開発を行っています. | 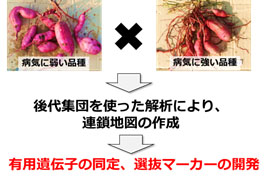 |
レトロトランスポゾン,次世代シーケンス,育種,品種判定,遺伝解析,そしてサツマイモ…etcに興味をおもちの方,ぜひ一度遊びに来てくださいね!サッカー好きな方、BBQ好きな方も大歓迎です!


研究業績リスト
・Monden, Y. and Tahara, M. (accepted) Plant transposable elements and their application to genetic analysis via high-throughput sequencing platform. The Horticulture Journal.
・Monden, Y., Hara, T., Okada, Y., Jahana, O., Kobayashi, A., Tabuchi, H, Onaga, S. and Tahara, M. (accepted) Construction of a linkage map based on retrotransposon insertion polymorphisms in sweet potato via high-throughput sequencing. Breeding Science.
・Monden, Y., Yamaguchi, K. and Tahara, M. (2014) Application of iPBS in high-throughput sequencing for the development of retrotransposon-based molecular markers. Current Plant Biology 1, 40-44.
・Monden, Y., Yamamoto, A., Shindo, A. and Tahara, M. (2014) Efficient DNA fingerprinting based on the targeted sequencing of active retrotransposon insertion sites using a bench-top high-throughput sequencing platform. DNA Research 21, 491-498.
・Monden, Y., Fujii, N., Yamaguchi, K., Ikeo, K., Nakazawa, Y., Waki, T., Hirashima, K., Uchimura, Y. and Tahara, M. (2014) Efficient screening of long terminal repeat retrotransposons that show high insertion polymorphism via high-throughput sequencing of the PBS site. Genome 57, 245-252.
・Monden, Y., Takasaki, K., Futo, S., Niwa, K., Kawase, M., Akitake, H. and Tahara, M. A rapid and enhanced DNA detection method for crop cultivar discrimination. Journal of Biotechnology 185, 57-62.
・Monden, Y., Takai, T. and Tahara, M. (2014) Characterization of a novel retrotransposon TriRe-1 using nullisomic-tetrasomic lines of hexaploid wheat. The Scientific Reports of the Faculty of Agriculture 103, 21-30.
・門田有希・高井健・田原誠・梅野佑太・中村遼太.(2014) 新規活性型レトロトランスポゾンTriRe-1の挿入多型を利用した、High-throughputなコムギ品種判定マーカーの開発. DNA多型 22, 60-65.
・Naito, K., Monden, Y., Yasuda, K., Saito, H. and Okumoto, Y. (2014) mPing: The bursting transposon. Breeding Science 64, 109-114.
・秋竹広翔・田原誠・門田有希・高崎一人・布藤聡.(2013) イチゴにおけるレトロトランスポゾン品種識別マーカーの開発. DNA多型 21, 64-72.
・門田有希・山本彩加・田原誠.(2013) 活性型レトロトランスポゾンを利用したアントシアニン含有紫サツマイモ品種識別用DNAマーカーの開発. DNA多型 21, 47-54.
・高井健・田原誠.(2012) コムギにおいて品種間挿入多型を示すレトロトランスポゾン配列の探索. DNA多型 20, 80-90.
卒業生・修了生進路
・ 大学院進学、公務員、全農、 一般企業(ロッテ、東洋ナッツ、山田養蜂場など)