“動く遺伝子”で遺伝解析を効率化~解析困難だった農作物にも利用可能~
2014年08月08日
岡山大学大学院環境生命科学研究科ゲノム遺伝解析学分野の田原誠教授、門田有希特任助教らの研究グループは、“動く遺伝子”と“次世代シーケンス”を利用し、効率的な遺伝解析技術を開発。サツマイモにおいて、品種間の遺伝的な関係を明らかにするとともに、品種判定マーカーを開発しました。
本手法は他の農作物にも応用可能です。サツマイモのように、これまで生物学的な理由で解析が困難であった農作物種において遺伝解析を加速化することが期待できます。これによって、美味しさや病害虫に強いなどの望ましい遺伝子を持つ系統を判定し、優れた農作物品種の作出を飛躍的に効率化することが期待されます。
<業 績> 本手法は他の農作物にも応用可能です。サツマイモのように、これまで生物学的な理由で解析が困難であった農作物種において遺伝解析を加速化することが期待できます。これによって、美味しさや病害虫に強いなどの望ましい遺伝子を持つ系統を判定し、優れた農作物品種の作出を飛躍的に効率化することが期待されます。
岡山大学大学院環境生命科学研究科ゲノム遺伝解析学分野の田原誠教授、門田有希特任助教、進藤彰子研究補助員、山本彩加(農学部4回生)らの研究グループは、サツマイモの“動く遺伝子”に注目し、 “次世代シーケンス”とよばれる技術を組み合わせることで効率的な遺伝解析を行いました。これにより、サツマイモ品種間の遺伝関係を明らかにし、また品種を特定するためのDNAマーカーの開発に成功しました。
本研究成果は、2014年6月16日にオンライン科学雑誌『DNA Research』(Oxford Journals) に掲載されました。また、米国のオンライン出版『GenomeWeb』にもインタビュー記事が掲載されました。
サツマイモは、他の農作物と比べて遺伝解析が非常に難しく、DNAマーカーの開発が遅れていました。その理由としては、サツマイモが倍数体(六倍体)であり、遺伝が複雑なためです。さらに、その全ゲノム配列情報もいまだに解読されていない、などが挙げられます。しかし、有用な品種を効率的に育成し、育成者の特許権を守るためには、遺伝情報を用いた解析手法の確立が必要不可欠でした。
そこで今回、研究グループは、レトロトランスポゾンとよばれる“動く遺伝子”に注目しました。これは、真核生物(植物・動物・菌類など)のゲノム中に存在し、自分のコピーを作って増えることができます。また、新しく作られたコピーは、一度DNA配列中に入ると安定して遺伝します。そのため、品種間で異なる場所にあるコピーは、DNAマーカーとして使うことができます。
また、研究グループは効率的な遺伝解析を行うため、近年急速に普及した“次世代シーケンス”を利用。短期間で数千万という非常に多くのDNA配列のデータを得ることができました。そして、解析結果からサツマイモ品種間の遺伝的な関係を明らかにし、すでに知られていた品種間の親子関係とほぼ一致することを確認。今まで困難であったサツマイモにおいて、得られた遺伝子データから品種管理ができることを実証しました。さらに、品種を特定するためのDNAマーカーの開発にも成功しました。これにより、より的確な品種の特定を行うことが出来るようになりました(図)。
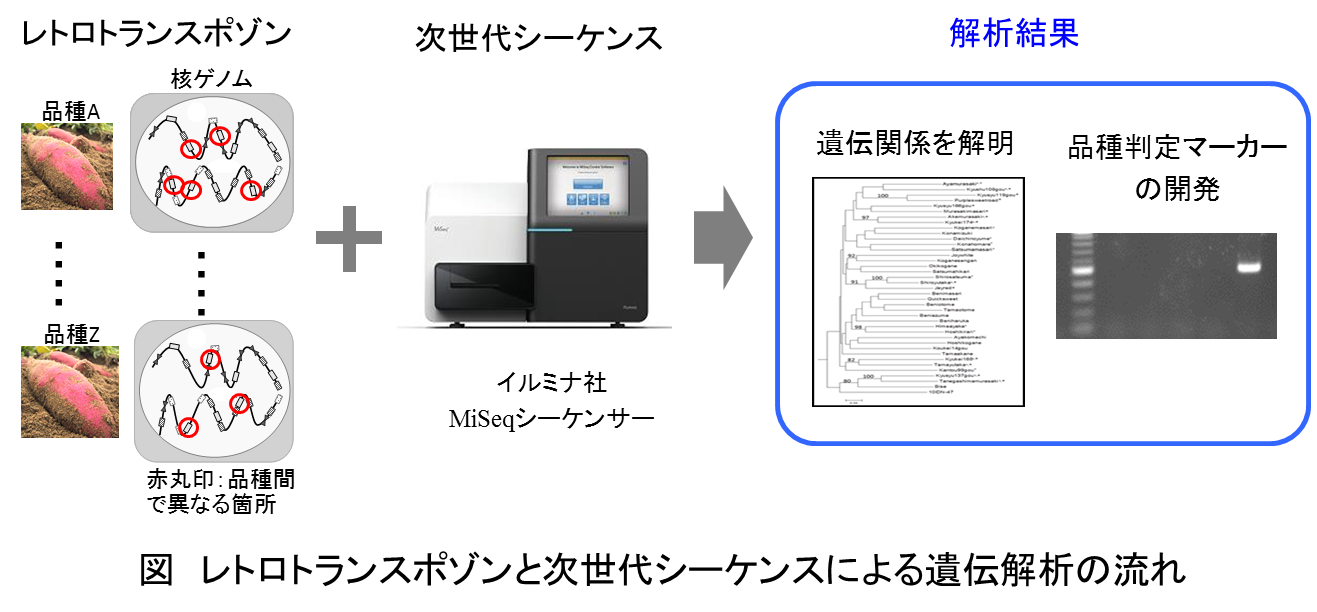
<見込まれる成果>
現在、さまざまな農作物において遺伝情報を用いた解析が行われています。しかし、農作物の中には、遺伝解析が難しく、あまり進んでいないものもあります。サツマイモもそのうちのひとつです。
“動く遺伝子”と“次世代シーケンス”用いた手法は、他の農作物にも利用することができます。そのため、未だに全ゲノム配列が解読されていない、あるいは解析が進んでいない農作物においても、農業上の有用な遺伝子を効率的に見つけ出すことが期待できます。
今後、より良い品種改良(病害に強い、おいしい、栄養価が高いetc)や、貴重なわが国の「日本品種を守る」ための技術を開発する取り組みがさらに活発になると期待されます。
<補 足>倍数体:2セットよりも多い染色体をもつ個体を示します。通常、受精後に母親と父親から1セットずつ、計2セットの染色体が受け継がれます(二倍体)。ヒトを含め、動物の多くは二倍体です。しかし、植物の中には、3セット(三倍体)、4セット(四倍体)など2セットよりも多い染色体をもつ倍数体が過半を占めます。例えば、ジャガイモは四倍体、コムギは六倍体です。
ゲノム配列の解読:その生物が持つすべてのDNAの塩基配列を明らかにすることです。ヒトのゲノムは2003年に解読終了の宣言がなされ、30億個もの塩基、A(アデニン)、G(グアニン)、T(チミン)、C(シトシン)のつながりの順番が明らかとなりました。DNAの塩基配列を読み取るシーケンサー技術の開発、発展により、現在さまざまな生物種のゲノム配列が解読されています。
レトロトランスポゾン:真核生物のゲノム中にある転移性DNA配列(トランスポゾン)の一種で、自身のコピー配列を作り増幅します。特に植物ゲノムにおいて占める割合が高いのが特徴です。ゲノム中には非常にたくさんのレトロトランスポゾンがありますが、大部分はゲノム進化の過程で、増幅能力を失っています。しかし、ごく稀に今でも増幅能力をもつレトロトランスポゾンがあることが知られています。
次世代シーケンス:近年急速に普及した、新しい原理に基づくシーケンサーによる解析を示します。従来のキャピラリーシーケンサーと比較し、解析能力が大幅に向上し、短時間で大量の配列データを出力可能になりました。遺伝学・ゲノム解析が飛躍的に進み、生命科学に“革命”を起こしたとも言われています。
本研究は、農林水産省の平成24~26年度「新たな農林水産政策を推進する実用技術開発事業」の助成を受け実施しました。
発表論文はこちらからご確認いただけます。
発表論文:Monden Y, Yamamoto A, Shindo A, Tahara M. Efficient DNA Fingerprinting Based on the Targeted Sequencing of Active Retrotransposon Insertion Sites Using a Bench-Top High-Throughput Sequencing Platform: DNA Research, 2014, (doi: 10.1093/dnares/dsu015)
報道発表資料はこちらをご覧ください
<お問い合わせ>
(所属)岡山大学大学院環境生命科学研究科
ゲノム遺伝解析学分野 教授
(氏名)田原 誠
(電話番号)086-251-8312
(FAX番号)086-251-8388